入门教程¶
主要讲述PyMOL中常见的图形界面(鼠标)操作
PyMOL软件界面介绍¶
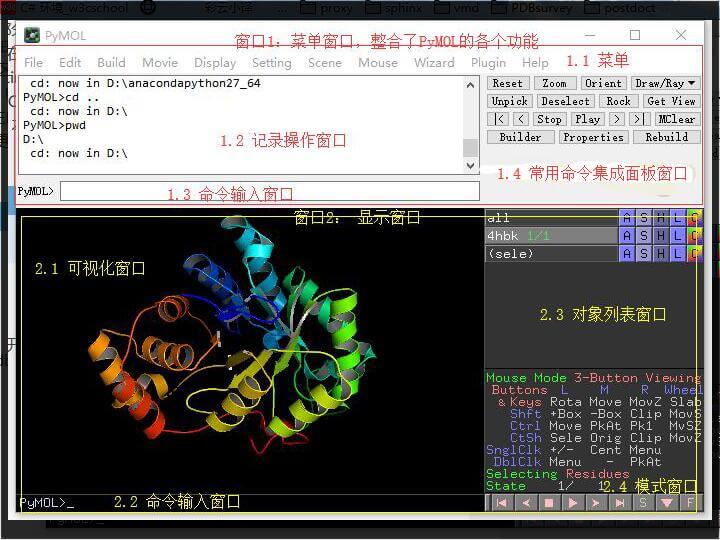
PyMOL 软件界面如下图所示,主要由2部分组成。第一部分是菜单窗口和第二部分是显示窗口。 PyMOL2 和PyMOL1一个明显区别: 在PyMOL2中,这2部分默认是一个整体,在PyMOL1中这2部分是分开的。 在Display->Toggle Floating 可以拆分这2个窗口,快捷键是Ctrl+E, 刚开始PyMOL的界面主要是通过tkinter实现的,在PyMOL2中逐渐采用PyQT技术实现。 和PyMOL1 相比,PyMOL2 表现能力更强,功能更多,本教程以PyMOL2为例进行讲解,大部分 内容也适用于PyMOL1.
窗口1:整合了PyMOL的各个功能,主要包含如下部分
1.1 菜单窗口 1.2 操作记录显示窗口 1.3 命令输入窗口 1.4 常用命令集成面板
窗口2:分子操作和显示窗口,主要包括如下部分:
2.1 可视化窗口 2.2 命令输入窗口 2.3 分子对象列表窗口 2.4 模式窗口
注解
本节教程是有顺序的。
窗口拆分和合并¶
在Display->Toggle Floating 可以拆分这2个窗口,快捷键是Ctrl+E,
PyMOL内置的超强可视化示例¶
文本教程¶
打开PyMOL,点击1.1菜单窗口的Wizard菜单,然后点击Demo->Representations 然后在2.3 对象窗口 和 2.4模式窗口之间会出现各种示例:
- Representations
- Cartoon Ribbons
- Roving Detail
- Roving Density
- Transparency
- Ray Tracing
- Sculpting
- Scripted Animation
- Electrostatics
- CGOs
- Molscript/R3D Input
- End Demonstration 关闭示例
从中我选择了几个示例图片展示下:
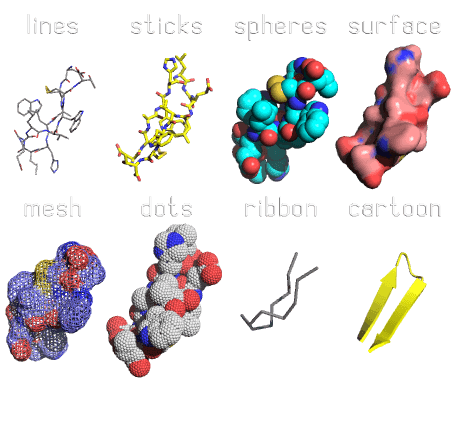
Fig 1. Representations |
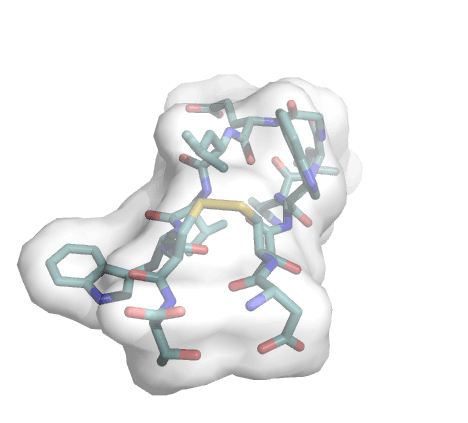
Fig 2. Transparency |

Fig 3. Electrostatics |
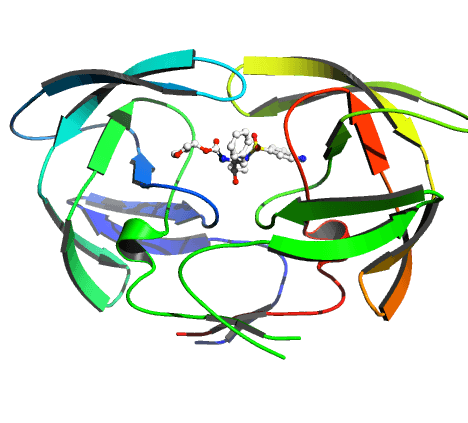
Fig 4. R3D |
从这些示例中,我们可以感受到PyMOL的强大表现能力。
视频教程¶
设置和查看工作路径¶
点击1.1菜单窗口 File->Working Directory->Change 可以查看现在的工作目录在哪里,也可以设置新的路径作为工作目录。
工作目录的作用:默认文件的打开和保存都是从该文件开始。下载文件的保存位置也是在工作目录。
工作目录设置习惯
- 建议: 1. 不同项目设置不同的工作目录
- 设置一个默认工作目录
- 不要把工作目录设置在软件安装目录
- 双击PDB文件打开PyMOL,会自动切换工作目录到该PDB文件所在目录。
- 从软件安装处打开PyMOL, 则工作目录为软件安装目录
这里我设置工作目录为 D:\PyMOLstartedManual
下载蛋白¶
从PDB网站上下载蛋白,如 4hbk.pdb, 也可以直接通过PyMOL 下载蛋白,点击菜单栏中的 File->Get PDB,如下图图所示, 在PDB ID 对应的框中填入PDB的编号就可以了,不要包含后缀.pdb 。
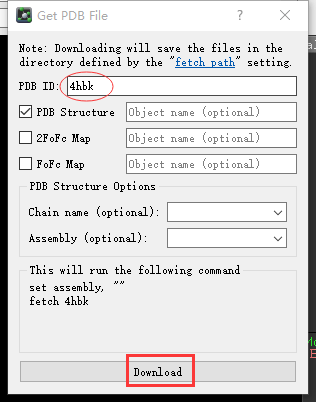
点击Download,你会发现PyMOL会自动加载该蛋白,在工作路径 D:\PyMOLstartedManual 下面出现 4hbk.cif 文件,随着PDB解析的结构越来越大,PDB格式文件的局限性就暴露出来,不能超过9999个原子,因此正在逐渐用cif 格式取代pdb 格式。
视频教程¶
蛋白结构的加载和展示
蛋白的展现形式(show)¶
文本教程¶
在2.3对象列表窗口中,我们可以看到现在有2个object 名字,
- 1个是all, all 不是真实的object,它代表了所有的object
- 1个是4hbk, 4hbk 就是我们刚刚载入的蛋白
每个object 都有对应的A S H L C操作,如下图所示:
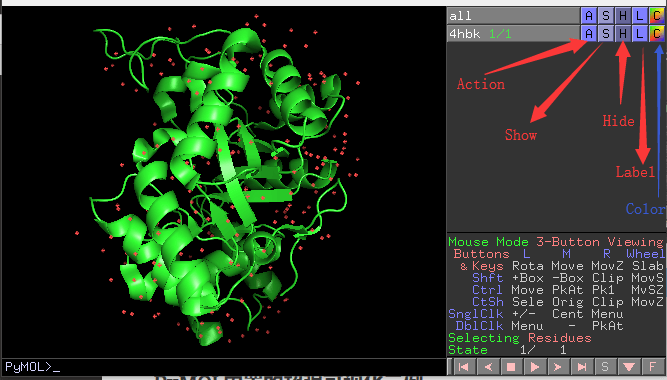
- Action 主要包含了对object的常用操作的集合,如 复制、删除object,对object加氢,展示Object等,
更多详情参见蛋白对象的Action操作。 - Show 将object 渲染成cartoon 、line、stick lines sphere surface mesh dots ribbon 等模式 - Hide 根据object的状态或者描述进行相应的掩藏 - Label 显示object中残基 原子等名称或者属性 - Color 对Object 进行着色
下面对Show 着重讲解: show 有2类操作方法:
- show as 分别点击S->as->cartoon 和S->as->stick,
- 我们可以观察到AS模式是把原有的渲染模式抹除后再重新渲染,经过上述操作后仅仅显示stick形式
- show 点击S->as->cartoon 再点击S->stick;
- 我们可以观察到SHOW方法,是保留原有的渲染,再添加新的渲染。
我们先对4hbk object点击 S->as->cartoon,然后点击S->as->stick,效果如图Fig5
我们先对4hbk object点击 S->as->cartoon,然后点击S->stick,效果如图Fig6
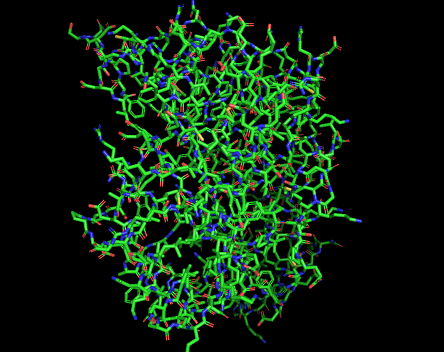
Fig 5. as stick |
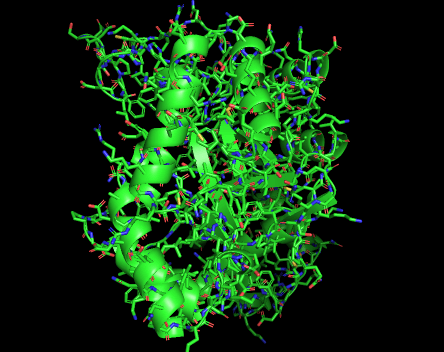
Fig 6. stick |
大家可以下载demo1的示例蛋白文件 demo1_pp.pdb,点此 pdb 下载。
然后在PyMOL中点击File -> Open-> demo1_pp.pdb 打开该文件。
- 点击 S->as->lines
- 点击 S->as->sticks
- 点击 S->as->spheres
- 点击 S->as->surface
- 点击 S->as->mesh
- 点击 S->as->dots
- 点击 S->as->ribbon
- 点击 S->as->cartoon
观察该分子对象的不同形态,如图1所示。
视频教程¶
蛋白对象的简单平移、旋转、缩放¶
首先将鼠标移动到2.1可视化窗口
- 平移,按住鼠标中键不放,然后上下左右移动,进行体会,蛋白会随着鼠标而移动
- 旋转,按住鼠标左键不放,然后上下左右移动鼠标,蛋白会进行旋转
- 缩放,按住鼠标右键不放,然后上下移动,蛋白会进行缩放
- 切割 滚动鼠标中键, 建议将蛋白渲染成surface模式,然后滚动鼠标中键
当软件不能正常使用上述操作,可以点击
- File->Reinitialize->Original Settings (推荐)
- File->Reinitialize->Everything 注意的是该操作会删除当前所有的Object,
蛋白对象的action操作¶
对于4HBK体系里面没有小分子,一些相互作用操作不方便演示,我会在在后面进行更详细的讲解。
第一部分: 常用显示操作
- 点击 A->preset->simple 显示蛋白的简单形式
- 点击 A->preset->ball and stick 显示球棍模型
效果如下图所示:
- 点击 A->preset->b-factor putty 基于bfactor数值显示蛋白的柔性
- 点击 A->preset->publication 高质量出版标准
第二部分 对象的操作
- 删除水分子 A->remove waters
- 增加删除氢原子
- 在line和stick 模式下面可以看到H原子,cartoon模式下面看不到氢原子 因此在line或者stick模式下,执行下述操作。
- A->Hydrogens->remove 删除所有氢原子
- A->Hydrogens->add 增加所有氢原子
- A->Hydrogens->remove non polar 删除所有非极性氢原子,我们可以看到C上的氢原子全部被删除
- A->Hydrogens->remove 再次删除所有氢原子
- A->Hydrogens->add polar 增加极性氢原子
第三部分 对象的复制(追加) 剪切 删除 重命名
- 复制 A->Copy to object->new
- 复制追加 (object1)A->Copy to object->
- 重命名 obj01-> 4hbk_02
- 对obj01 点击A->rename object
- 删除 A->delete object
- 剪切 适合选中的对象 A->Extract
我们可以通过剪切的操作把配体和蛋白分开,首先选中配体,然后点击A->extract object 就可以了, 原来结构中的配体就跑到obj01中了,这样就分开了蛋白和配体。
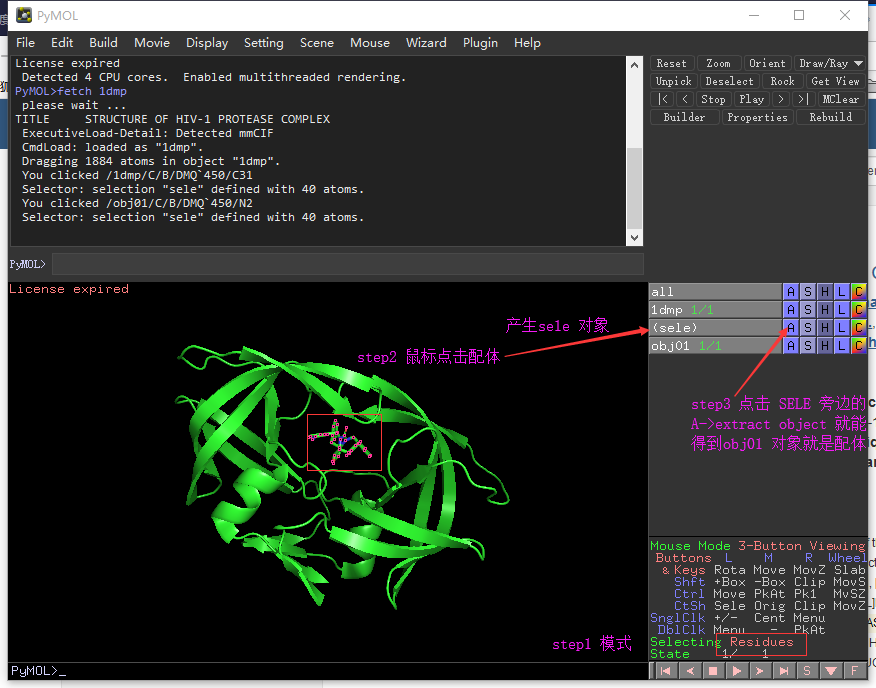
第四部分 Action->generate 操作
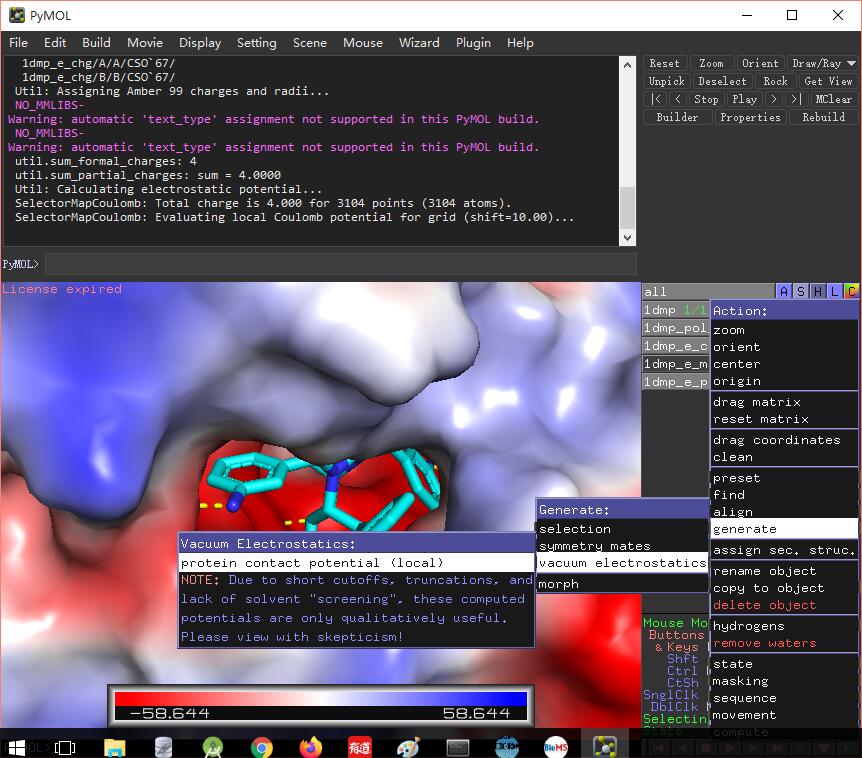
- 显示蛋白的静电势图 Action->generate->vacuum_electrostatics->protein contact potential 就可以了
查看小分子和蛋白的氢键作用¶
由于4hbk 蛋白中没有小分子,这里我以 PDB id: 为例演示。 点击 A->preset->ligand sites 效果如图所示,其中黄色的虚线就是氢键。
对标注的氢键,查看距离和角度,进一步确定氢键的合理性和强度。
查看小分子和蛋白的相互作用,推荐软件 schrodinger中ligand interaction。 制作相互作用的二维图,根据提示,然后再在pymol确认并绘制这些相互作用。
查看受体和配体之间的极性作用¶
鼠标选中配体,出现“sele” object,然后点击action->find polar contacts->to other atom in object.
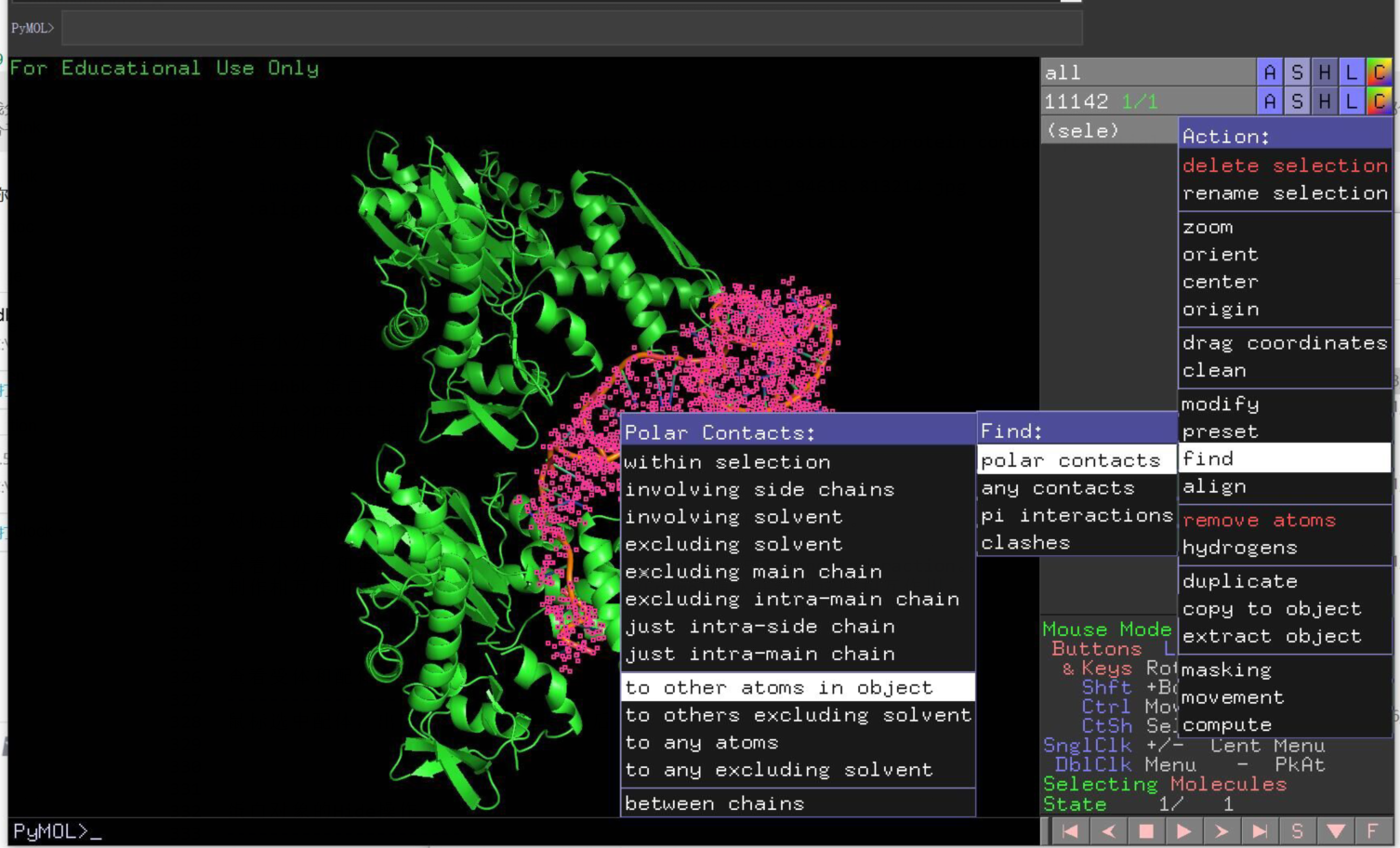
注解
to other atom in object: 受体和配体之间的相互作用
to any atom: 同时考虑受体和配体之间的作用,以及配体内的极性作用。
蛋白对象的Hide操作¶
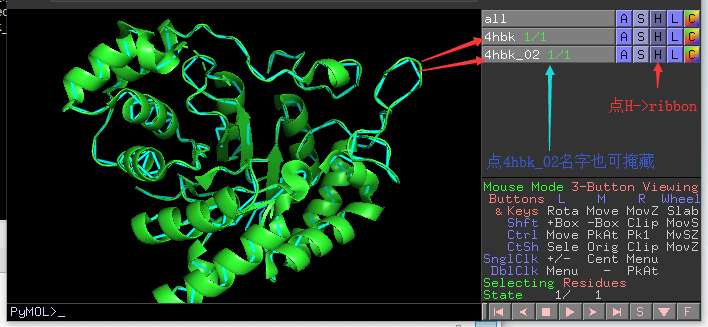
和remove delete 操作相比,hide操作更加温和, 把不需要的东西暂时掩藏起来,通过Show 可以重现显示出来。 我们先上述方法,构建4hbk 和4hbk_02 两个object, 这一次把4hbk 设置成cyan 颜色的cartoon, 4hbk_02设置成green颜色的ribbon,如图所示。 如果要掩藏4hbk_02,有2种方法 - 对4hbk_02 点击H->ribbOn - 直接点击4hbk_02的名字
选择蛋白的特定残基¶
我们通过软件 D3Pockets 可以确定4hbk中口袋中氨基酸残基的组成:
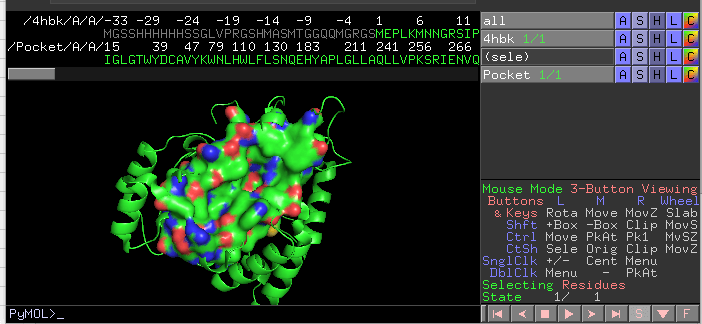
选择感兴趣的object周围4A的氨基酸残基¶
- step1. 鼠标点击感兴趣的object,在右侧窗口出现sele的object
- step2. 点击 sele 旁边的A(action)->modify ->around ->residues within 4A
蛋白对象的着色操作¶
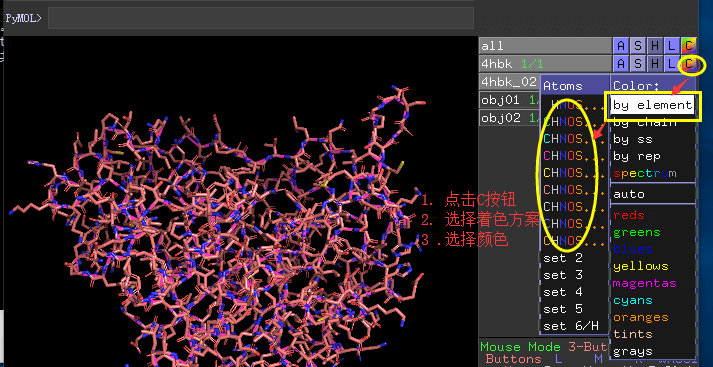
pymol中内置了多种不同的着色方案,如图所示:
按照原子类型着色
点击object上的 C按钮 -> by element
按照二级结构着色
按照b-factor着色
按照整体着色
对象的Label操作¶
添加label¶
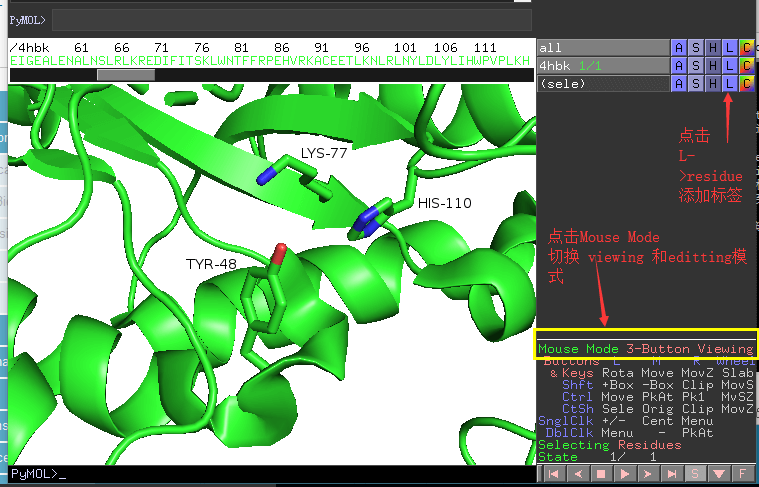
选择需要Label的对象 | 然后点击对象上的L按钮,根据需要,可以标记残基的名字,原子的名字,范德华半径、元素的名字等。
对于蛋白醛糖还原酶(PDB ID: 4HBK),我们在UniProt 数据库中查询得到,其口袋中的重要残基有: TYR48、LYS77 和 HIS110。
我们对这三个残基展示其为stick模式,并掩藏主链,并通过label按钮标注其氨基酸残基的名字。 移动Label,使其更加清晰可见。具体操作流程如下:
效果如下图所示:
移动标签label¶
以PDB ID: 1w22蛋白为例,为其中的锌离子添加标签。
注意
注解
sphere模式下不能添加label;借用PS中复制图层的概念。为锌离子添加label。
操作如下面的动画所示。
设置label的样式¶
调整配体的位置¶
pymol中包含配体和蛋白,如何调整配体和蛋白的相对位置。
以PDB ID: 1hkv 为例,其中配体小分子为PLP
操作如下面的动画所示。
调整修改二面角¶
- step1 Mouse Mode调整到edit模式;
- step2 按住ctrl+鼠标右键 点击需要修改的二面角的中间键,需要转动左边,就点击左边。需要转动右边,就点击右边。
- step3 按住ctrl+鼠标左键,上下移动鼠标,调整二面角;
- step4 调整到想要的二面角,Mouse Mode调整到view模式;
测量距离¶
点击菜单栏中的Wizard->Measurement 就可以进行距离测量。然后分别选择两个2个原子就可以显示这2个原子的距离。
比如我们想测定TYR48中侧链上的O原子到Lys77侧链N原子的距离,如下操作即可:
效果如图所示:
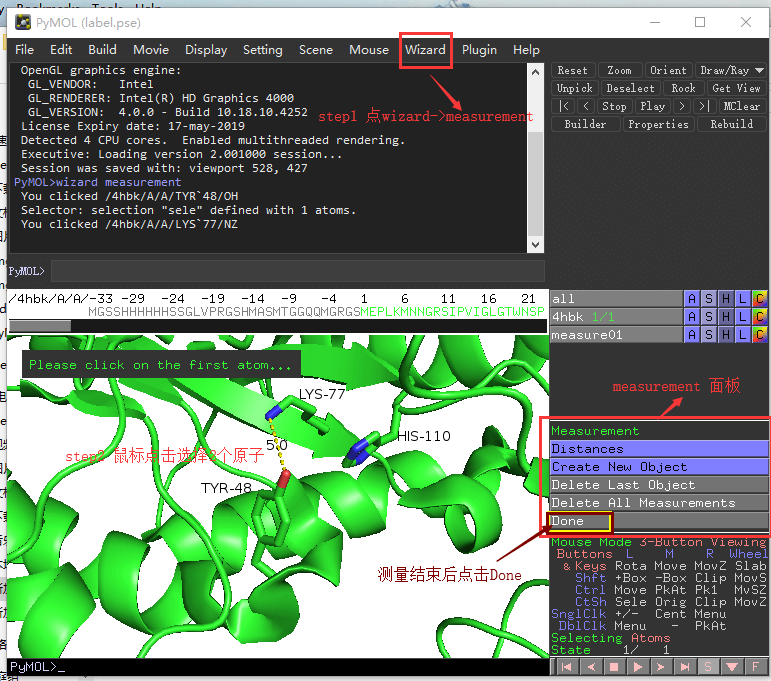
作业:
1. 测定TYR48的α碳原子和LYS77的α碳原子的距离? 答案是11.6A
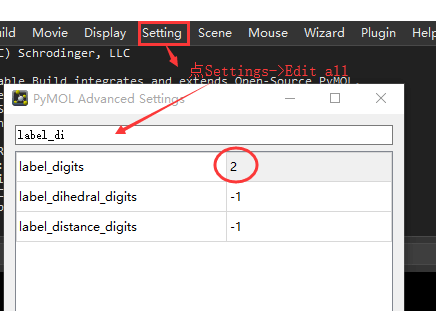
默认测定的距离是保留1位小数,如果想要显示2位小数,可点击菜单栏中的Setting->Edit All, 然后搜索label_digits,找到该索引,并把后面的数值设置为2就可以了。如图所示:
测量角度¶
比如我们想测定Lys77侧链CE、NZ原子和的TYR48中侧链上的O原子的角度,如下操作即可:
效果如图所示:
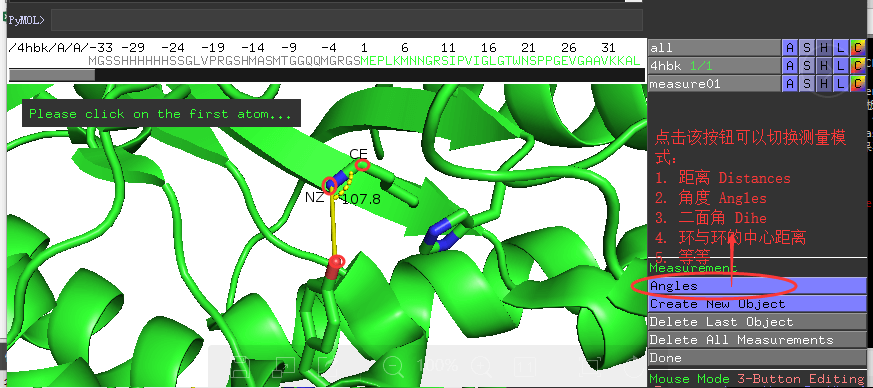
测量二面角¶
同测量距离和角度的操作,只需要将测量模式切换为Dihedrals。
测量环的距离¶
同测量距离和角度的操作,只需要将测量模式切换为Dihedrals。
设置透明度¶
鼠标操作只能基于显示模式进行设置,如:cartoon surface sphere stick等
除了可以设置透明度外,还可以设置透明的方式,有如下几种透明模式:
在Label操作中,我们将整个蛋白展示为cartoon,并将HIS 等3个残基展示为stick模式; 这时候我们设置cartoon为透明50%,用4种不同的透明模式渲染,效果如下:
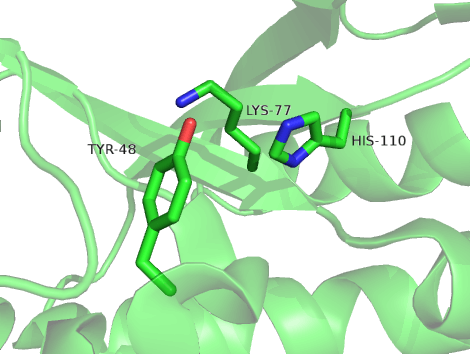
Fig 1. Uni-layer 透明效果 |
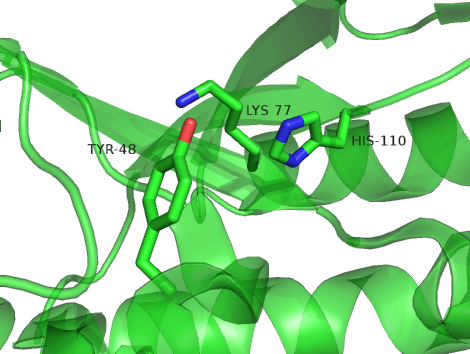
Fig 2. Multi-Layer 透明效果 |
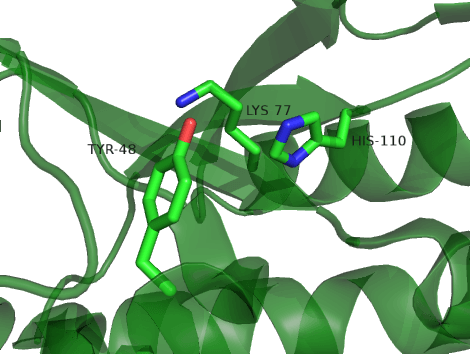
Fig 3. Multi-Layer(real time oit) |
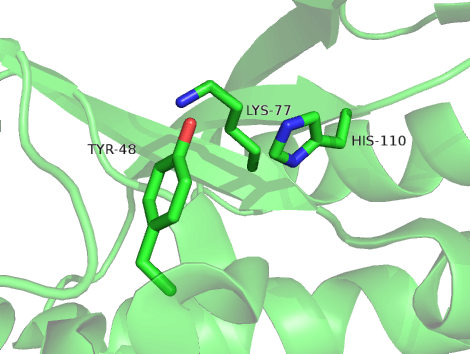
Fig 4. Fast-ugly |
雾化(fogging)处理¶
PyMOL中支持各种雾化处理,保证depth_cue是开启的。
前面清晰,后面雾化可通过滚动鼠标中键进行调节。
向上滚动,雾化程度减轻;向上滚动,雾化程度加深。
Fig 1. fogging 效果 |
Fig 2. unfogging 效果 |
完全不想要雾化处理,可以点击 Display->Depth cue (Fogging) 取消。
或者使用命令关闭。
set depth_cue, 0
设置不同的光照模式¶
PyMOL2 中内置5种不同的光照模式:default, metal(金属), plastic(塑料), rubber(橡胶), X-ray。
点击Plugin->lighting Settings进行设置不同的光照,如下图所示图。
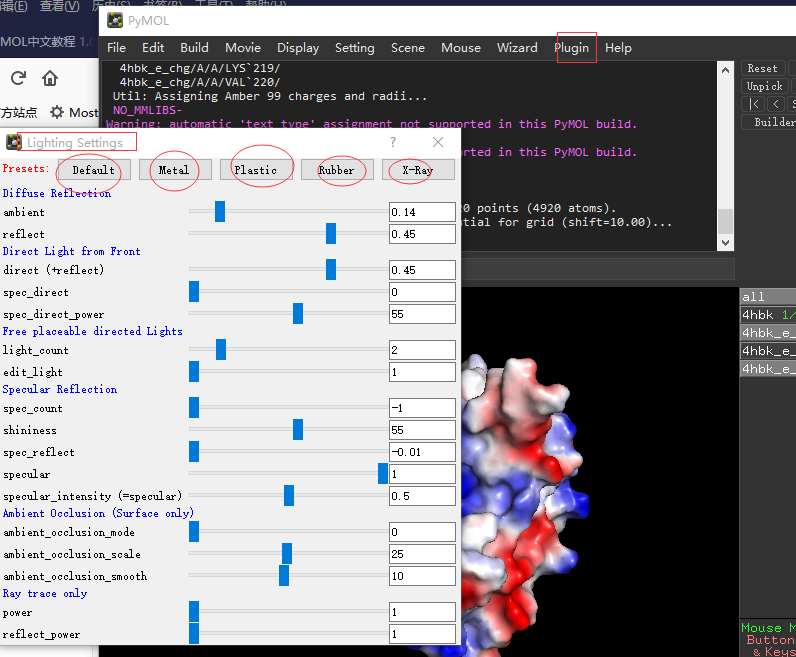
除了默认的5种模式外,你也可以通过设置光源的参数,达到自己想要的效果。
下面,我们查看蛋白的静电势表面在不同光照模式下的效果。

Fig 1. Default光照效果 |

Fig 2. Metal光照效果 |

Fig 3. Rubber光照效果 |

Fig 4. Xray光照效果 |
选择模式:¶
根据不同的需求,切换到不同的选择模式,快速选择自己想要的原子:
我们先将蛋白Show->as wire;然后把MET-1第一个氨基酸残基show stick;如图所示,
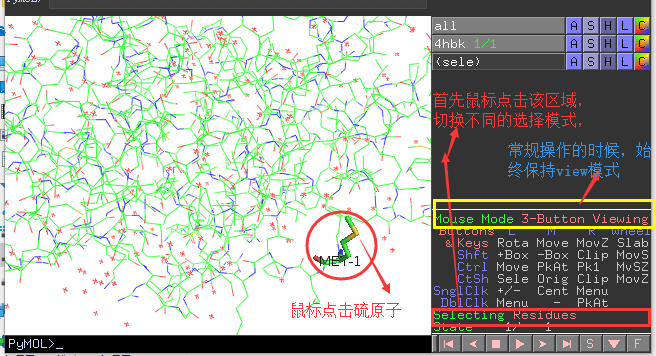
我们切换到不同的模式,选择甲硫氨酸上的硫原子。
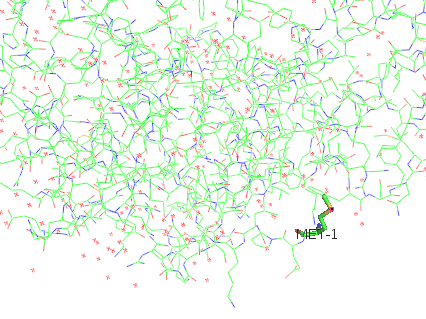
Fig 1. atoms模式下点击硫原子的效果 |
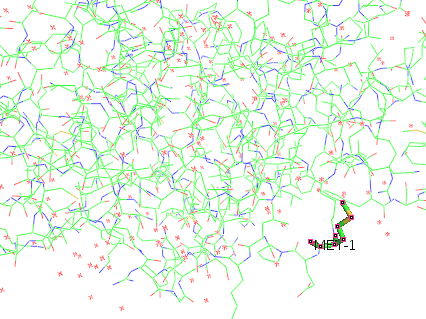
Fig 2. res模式下点击硫原子 |
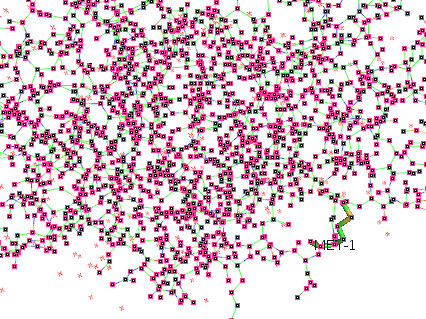
Fig 3. chain模式下点击硫原子 |
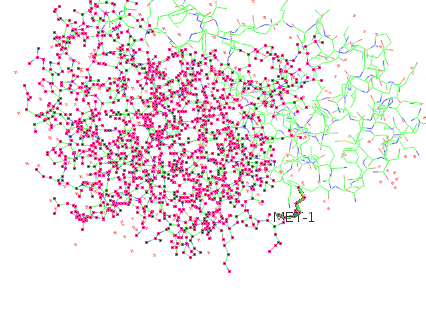
Fig 4. mol模式下点击硫原子 |
从上图我们也可以看到,不同模式的区别。这里我简单解释下:
保存或者导出结果¶
PyMOL 和 PhotoShop 有点类似,PyMOL中的Object 类似于 PhotoShop中的图层。
- PyMOL 可以将会话保存为pse文件,File->Save Session;pse 文件类似于PS中的psd文件,方便修改调整。
- PyMOL 可以将object 导出为结构文件,File->export molecule,然后从selection的下拉框中选择需要导出的object, all 代表所有的object; enable 代表的可见的object;
- 保存图片,File->export image as->png; 在新版本的,可以使用右上角的Ray/Trace按钮,设置图片大小 分辨率,进行保存图片。
4. 保存动画,File->export movie as->mpeg; PyMOL仅仅内置了 mpeg_encode 编码视频方式,默认只能保存mpg格式的动画。 可自行下载ffmpeg,从而可以保存为多种格式,如gif,mov,mpg等
设置pse的版本号,建议版本为0.99, 这样PyMOL 和 PyMOL2都可以打开pse 文件
set export_pse_version,0.99
删除蛋白的一条链,然后保存为pdb¶
step1. 切换到chain模式
step2. 点击需要需要删除的Chain就可以选中chain
step3. 对新产生的sele object进行删除。
移动object在列表中的顺序¶
鼠标右击待移动的object,按住鼠标不放,移动鼠标,调整object在列表中顺序。