进阶教程¶
主要讲述如何用PyMOL 绘制精美图片的案例,以及制作动画。
注解
动画更加形象生动,做PPT的时候加入一些动画,更能引起听众的兴趣和注意力!
注解
使用图片帧的方式制作动画的时候,保存的图片背景不能是透明的,否则会出现重影的现象。 默认导出图片背景是透明的,点击右上角 draw/ray, 取消勾选 transparent。
简单规则¶
对于缺少美感的人来说,如何绘制精美图片,有如下规则和方法: 1. 参考文献中图片,进行绘制 2. 文献中蛋白的颜色通常设置为灰白色。 3. 氢原子往往掩藏,尤其是非极性氢原子。
如何制作动画,有如下规则: 1. 平时注意积累动画库 2. 模仿别人制作的动画
安装ffmpeg方便直接导出mp4 和 gif 格式¶
安装方法一:
- step1 到 ffmpeg官网 下载ffmpeg,这里我以 windows为例 。
- step2 解压文件,将bin文件下的所有exe 程序拷贝的PATH路径下,或者将bin路径添加到PATH 环境变量中。
- step3 打开PyMOL,File->save move movie 选择ffmpeg 就可以保存动画。
安装方法2:
>>> conda install -c menpo ffmpeg
制作分子对接示例动画¶
- 准备材料(选定蛋白体系,配体)
- 分子对接,保留制作动画用的配体小分子构象
- 制作动画
3.1 导入蛋白,
3.2 通过命令导入小分子,并设置object的名字为lig
load conf1name.mol2, lig load conf2name.mol2,lig ... ...3.3 美化,调整角度,颜色,显示模式,动画速度等
3.4 mplay 播放动画
3.5 导出动画 ;
file->export movie as -> 选择ffmpeg/gif 保存动画为gif格式 或者 选择ffmpeg/mpeg4 保存动画为mp4 格式。
mp4格式的文件更小
这里我以蛋白 1ki2 为例制作动画,免费下载 素材 。
素材包含: 1. 蛋白构象;2. 小分子10个构象;3. mov.pml
打开pymol,执行如下命令,就可以创建动画了
load 1ki2_rec.pdb,rec
load ligconf01.mol2,lig
load ligconf02.mol2,lig
load ligconf03.mol2,lig
load ligconf04.mol2,lig
load ligconf05.mol2,lig
load ligconf06.mol2,lig
load ligconf07.mol2,lig
load ligconf08.mol2,lig
load ligconf09.mol2,lig
load ligconf10.mol2,lig
set movie_fps, 3
mplay
或者将上述命令写入到mov.pml 文件中,双击mov.pml 就可以。
我制作的动画如下

注解
pymol直接制作gif动画,会有黄色边缘。建议通过PyMol导出图片, 然后借助 ScreenToGif 将图片转换成gif动画。 流程如下:
- 打开ScreenToGif,选择编辑器模式;
- 点击文中中的媒体或项目,选中所有的帧图片上传到软件中;
- 点击编辑标签,选中所有图片,在重新设置中,设置每一帧的延时时间
- 最后点击文件,另存为gif文件就可以了。
制作小分子进入蛋白口袋的动画¶
这次我主要通过保存关键的frame 进行制作。
首先我们在脑海中构建几个关键图像,
1. 蛋白是空的。
2. 在蛋白外部出现小分子
4. 小分子慢慢进入口袋
帧和帧之间PYMOL会自动平滑过渡。 设置所有的帧的数目: mset 1 x300;
设置第一帧的图像,frame 1; 调整得到自己想要的视角;mview store;
在frame 100设置第二副图像,frame 100; 调整得到自己想要的视角;mview store;
在frame 200设置第二副图像,frame 200; 调整得到自己想要的视角;mview store;
mplay 播放动画。
注意: 这种方法不能改变蛋白的形态。
蛋白的口袋¶
蛋白构象变化¶
这里以腺苷酸激酶 (ADENYLATE KINASE)为例,PDB ID: 1AKE是关闭(close)构象,PDB ID: 4AKE是开放构象, 如下图所示
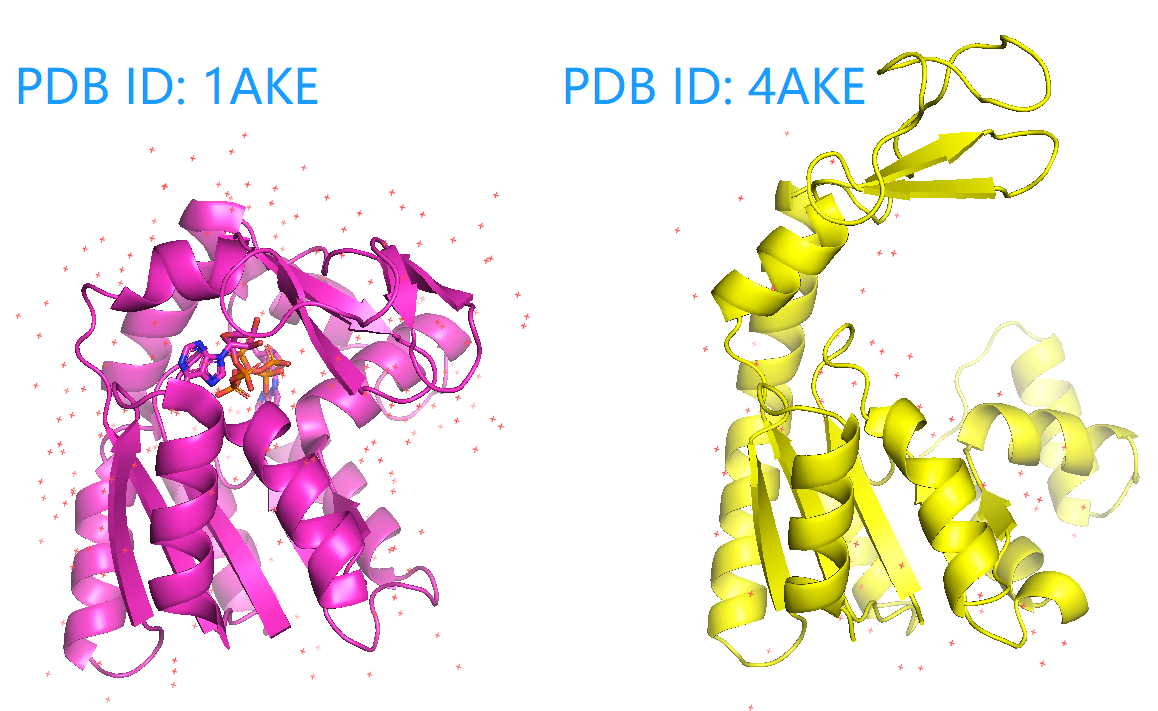
Schrodinger版本的PyMOL中内置了morph命令,开源版本的版本中没有morph命令。 给定两个或多个构象,morph命令可以创建从第一个构象到第二个构象的内插轨迹,平滑过渡。
命令版本:
fetch 1akeA 4akeA, async=0
align 1akeA, 4akeA
morph mout, 1akeA, 4akeA
鼠标点击版本
首先在Pymol中载入1ake_CHAINA和4ake_CHAINA;
然后进行align叠合;
最后在1ake的object上面点击A->generate->morph->to molecule->4ake就可以了。
动画效果如下,点我查看动画:
观察上面的动画,我们发现其是关闭构象缓慢打开到开放构象,然后直接跳跃到关闭构象了。 现在我们借助于morph构建1AKE是关闭(close)构象变化到PDB ID: 4AKE是开放构象再变化到1AKE是关闭(close)构象 的一个周期循环动画。
首先要确保1AKE和4AKE的残基存在原子对应关系,这里我提取A链并且删除侧链。也可以通过同源模建的方法进行准备。
PyMOL>load 1akeA.pdb,AKE
PyMOL>load 4akeA.pdb,AKE
PyMOL>load 4akeA.pdb,AKE
PyMOL>morph mout,AKE,AKE,0
动画效果如下,点我查看动画:

多构象的动画切换方法,首先进行预处理,保证构象中的原子有对应关系,然后把这些构象load到一个object中, 接着可以通过命令morph mout,AKE,AKE,0 或者鼠标操作A(action)->generate->morph->state->1-2-3-4-5就可以了。
这里我以腺苷酸激酶 (ADENYLATE KINASE)蛋白为例,准备了以下5个构象state:
- 构象1 初始close构象
- 构象2 初始open构象
- 构象3 和构象1 相同,初始close构象
- 构象4 初始close构象旋转90度(通过movement控制和rotate y,90,xxx命令)得到
- 构象5 初始open构象和构象4叠合后的构象。
动画效果如下所示, 点我查看动画 :

